Zmniejszanie wymiarowości przestrzeni poprzez ekstrakcję cech¶
W przypadku ekstrakcji cech dokonujemy pewnej transformacji wejściowego wektora atrybutów w wektor o mniejszej wymiarowości. W efekcie generujemy nowe cechy, przekształcone w stosunku do cech wejściowych cechy. Właściwej transformacji poszukuje się poprzez optymalizację różnych funkcji kryterialnych, zarówno liniowych jak i nieliniowych. W zależności od przyjętego kryterium można otrzymać cechy najbardziej reprezentatywne, czyli takie, które w sensie przyjętego kryterium najbardziej aproksymują dane wejściowe w przestrzeni o mniejszej wymiarowości, albo cechy, które zapewniają najlepszą separację pomiędzy klasami. Jednym z najbardziej znanych przykładów takich algorytmów jest Metoda Komponentów Głównych (PCA), przekształcająca w sposób liniowy dany zbiór wejściowy. Współczynniki tego przekształcenia określone są poprzez wektory własne odpowiadające największym wartościom własnym macierzy kowariancji z próby. Inną popularną metodą jest Liniowa Dyskryminacja Fisher (FLD). Celem tej metody jest wyznaczenie takiej prostej, aby po rzutowaniu obiektów wejściowych na tą prostą uzyskać najlepsze rozdzielenie pomiędzy klasami.
Liniowa Analiza Dyskryminacyjna¶
Metoda Liniowej Analizy Dyskryminacyjnej (LDA) (ang. Linear Discriminant Analysis) (por. [Hestie2001], [Fukunaga1990], [Duda2000], [Marques2001]) jest najstarszym podejściem do redukcji przestrzeni wymiaru cech. Podejście to zostało zaproponowane w 1936r. przez sir Rolanda Fishera [Fisher1936]. W metodzie tej zakładamy, że wektory obserwacji są wektorami z \(p\)-wymiarowej przestrzeni euklidesowej. Metoda LDA prowadzi do otrzymania reguły decyzyjnej opartej na funkcji liniowej. Autor metody rozważał jedynie przypadek z dwiema klasami, jednak jego rozwiązanie możemy uogólnić na przypadek większej liczby klas. Uogólnienie to przedstawimy w następnym podrozdziale, natomiast teraz rozważmy problem dwuklasowy.
Załóżmy, że mamy \(n\) próbek o znanej przynależności do klas, z czego \(n_1\) próbek należy do klasy \(C_1\), natomiast \(n_2\) próbek należy do klasy \(C_2\). Oczywiście \(n_1+n_2=n\). Niech zatem \(x_1,\ldots,x_n\) będą naszymi próbkami w \(p\)-wymiarowej przestrzeni cech \(X\).
Oznaczmy przez \(y_i\)
liniową kombinację cech \(x_i\). Występujący w powyższym równaniu \(p\) wymiarowy wektor \(w\) możemy traktować jako linię w \(p\) wymiarowej przestrzeni cech, natomiast \(y_i\) jako projekcję wektora \(x_i\) na tą prostą.
Ponadto niech
będzie pewną funkcją liniową. Załóżmy, że wartość funkcji \(h(x_i)>0\) jeśli element \(x_i\) należy do klasy \(C_1\), w przeciwnym wypadku obiekt \(h(x_i)<0\). Naszym celem jest znalezienie optymalnych współczynników \(w\) oraz progu \(w_0\). Zmienna
w rzutowanej jednowymiarowej przestrzeni jest klasyfikowana do jednej z klas w zależności od wartości. Jeśli \(y<-w_0\) to zmienna \(y\) zostanie zaklasyfikowana do klasy \(C_1\), natomiast jeśli \(y>w_0\) to \(y\in C_2\).
Ponadto jeśli zmienna \(x_i\) ma rozkład normalny, to \(h(x_i)\) także ma rozkład normalny oraz zachodzą warunki
gdzie funkcje \(\eta_i\) oraz \(\sigma_i^2\) są kryteriami miary separowalności klas w rzutowanej przestrzeni. Jeśli natomiast \(x_i\) nie ma rozkładu normalnego, wówczas z twierdzeń granicznych wynika, że \(h(x_i)\) nadal może mieć rozkład normalny dla wystarczająco dużego \(p\). Wartość oczekiwana \(h(x_i)\) wyraża się następującym wzorem
gdzie \(M_i\) jest wektorem wartości średnich w klasie \(C_i\). Wariancja \(h(x_i)\) jest postaci
gdzie \(S_i\) jest próbkową macierzą kowariancji dla klasy \(C_i\) daną wzorem
Oznaczmy przez \(J(\eta_1,\eta_2,\sigma_1^2,\sigma_2^2)\) funkcję kryterialną optymalizowaną w celu obliczenia wartości \(w\) oraz \(w_0\). Korzystając z twierdzenia o pochodnej funkcji złożonej, otrzymujemy
oraz
Ponadto,
Dokonując odpowiednich podstawień w (1) i (2) dostajemy
i
Przyrównując obydwie powyższe pochodne do zera otrzymujemy
oraz
Błąd w rzutowanej przestrzeni zależy tylko od kierunku wektora \(w\) a nie od jego długości. Dlatego też dla uproszczenia możemy usunąć wszystkie stałe, przez które mnożymy wektor \(w\). Stąd mamy wzór na postać optymalnych współczynników wektora \(w\)
gdzie
W zależności od wyboru funkcji \(J\) otrzymujemy różne postacie algorytmu Liniowej Analizy Dyskryminacyjnej. Jedną z możliwych postaci funkcji \(J\) jest funkcja kryterialna Fishera. Sprowadza ona algorytm LDA do algorytmu Liniowej Dyskryminacji Fishera (ang. Fisher Linear Discriminat) FLD.
Liniowa Dyskryminacja Fishera¶
Rozważmy funkcję kryterialną Fishera daną wzorem
Istotą liniowej dyskryminacji Fishera (ang. Fisher Linear Discriminant (FLD)) (por. [Hestie2001], [Fukunaga1990], [Duda2000], [Marques2001]) jest znalezienie takiej prostej \(w\), aby po rzutowaniu wektorów wejściowych na tą prostą uzyskać najlepsze rozdzielenie wektorów z klasy \(C_1\) oraz \(C_2\). Miarą rozdzielności klas przyjętą przez Fishera jest funkcja kryterialna (5). Istnieją różne metody pozwalające wyznaczyć wektor \(w\) maksymalizujący kryterium (5). Zgodnie z metodą opisaną w [Schalkoff1992] w tym celu wystarczy obliczyć pochodne funkcji \(J\) względem \(\sigma_1^2\) oraz \(\sigma_2^2\). Są one następującej postaci
Stąd dzięki (3) i (4) dla funkcji \(J\) postaci (5) dostajemy
oraz
Ponieważ w różnicy \(\eta_2-\eta_1\) nie występuje czynnik \(w_0\), kryterium Fishera nie zależy od progu \(w_0\). Czynnik ten zmienia jedynie długość wektora, który wyznacza kierunek prostej. Mając dany wektor \(w\) możemy dokonać klasyfikacji. Wówczas klasyfikowany wektor \(x_i\) oraz wektory wartości średnich w obu klasach rzutujemy na prostą wyznaczoną przez wektor \(w\). Następnie wyznaczamy odległości pomiędzy rzutem wektora \(x_i\) na prostą \(w\) oraz rzutami wartości średnich na tą prostą. Klasyfikowany wektor \(x_i\) przypisujemy do klasy, dla której obliczona odległość będzie mniejsza. Poniżej przedstawiamy schemat algorytmu FLD.
Algorytm 5 Algorytm liniowej dyskryminacji Fishera (FLD)
Dane: \(X_{train}\) - zbiór uczący, \(X_{test}\) - zbiór testowy, \(C_{train}=\{C_1,C_2\}=\{-1,1\}\) - etykiety klas dla zbioru uczącego
Wyniki: \(C_{test}\) - wektor przynależności do klas, \(w\) - wektor współczynników
- \(\eta_1=\frac{1}{n_1}\sum_{i=1}^{n_1}x_i^1 \quad \text{dla} \quad (x_i^1,C_1)\in X_{train}\times C_1\).
- \(\eta_2=\frac{1}{n_2}\sum_{i=1}^{n_2}x_i^2 \quad \text{dla} \quad (x_i^2,C_2)\in X_{train}\times C_2\).
- \(\sigma_1^2=\frac{1}{n_1}\sum_{i=1}^{n_1}(x_i^1-\bar{x}_i^1)(x_i^1-\bar{x}_i^1)^T \quad \text{dla} \quad (x_i^1,C_1)\in X_{train}\times C_1\).
- \(\sigma_2^2=\frac{1}{n_2}\sum_{i=1}^{n_2}(x_i^2-\bar{x}_i^2)(x_i^2-\bar{x}_i^2)^T \quad \text{dla} \quad (x_i^2,C_2)\in X_{train}\times C_2\).
- \(w=\left[\frac{1}{2}\sigma_1^2+\frac{1}{2}\sigma_2^2\right]^{-1}(\eta_2-\eta_1)\).
- Obliczanie wektora przynależności do klas \(C_{test}=\mathop{\rm sgn}{\left(w^T\cdot X_{test}\right)}\)
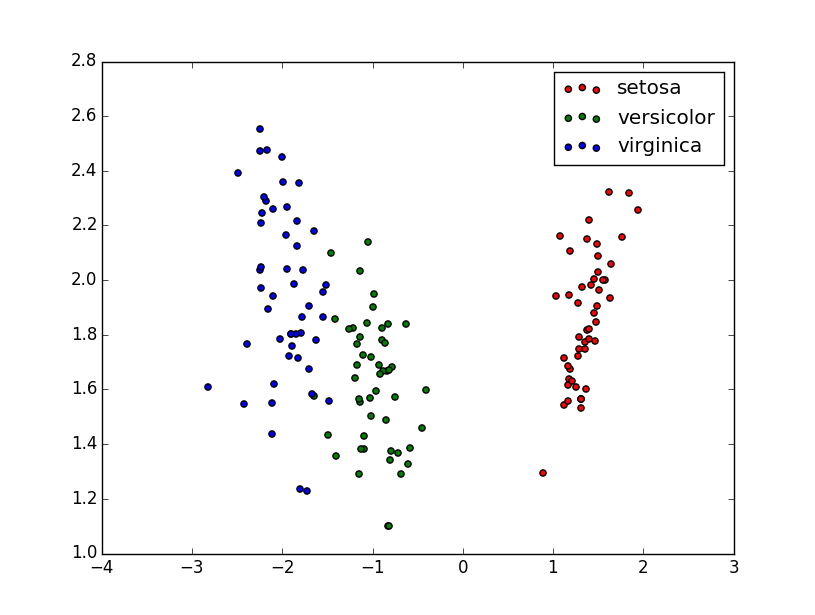
Rysunek 1 ilustruje prostą dyskryminacyjną oraz rzuty obiektów na prostą wyznaczoną przez wektor \(w\).
Problem wielu klas¶
Metodę Fishera dla dwu klas można uogólnić na przypadek większej liczby klas. Jako pierwsi uogólnienie to podali C. R. Rao w 1948 [Rao1948] oraz J. G. Bryan w 1951 [Bryan1951]. Zauważmy, że
gdzie \(\eta\) jest wektorem średnim dla wszystkich obserwacji. Macierz
możemy uznać za miarę zmienności międzygrupowej. Mówimy wówczas o macierzy kowariancji międzygrupowej. Powyższą macierz oznaczamy symbolem \(S_B\), tzn.
Ponadto, niech \(S_W\) oznacza macierz postaci
gdzie \(p_i\) jest prawdopodobieństwem a priori \(i\)-tej klasy, natomiast \(S_i\) oznaczają próbkową macierz kowariancji w klasie \(i\). Stąd na podstawie (6) i (7) kryterium Fishera (5) możemy zapisać w następującej, równoważnej, postaci
Uogólnienie algorytmu FLD dla większej liczby klas sprowadza się więc do uogólnienia wyrażeń (6) i (7). Wówczas wyrażenie (6) dla liczby klas \(L>2\) jest postaci
natomiast wyrażenie (7) przyjmuje postać
gdzie \(S_i\) jest macierzą rozproszenia wewnątrzklasowego dla \(i\)-tej klasy. Otrzymujemy w ten sposób uogólnioną liniową dyskryminację Fishera dla liczby klas \(L>2\).
Metodami opartymi na kryterium Fishera zajmował się między innymi W. Malina [Malina1981], [Malina1987], [Malina2001], [Kolakowska2005], a także Okada i Tomita [Okada1984].
Przykład 9
Rozważmy zbiór danych Iris. W zbiorze znajduje się 150 obiektów z trzech różnych gatunków irysów, a każdy z nich jest charakteryzowany przez cztery cechy: długość (w \(cm\).) listka kielicha kwiatowego, szerokość (w \(cm\)) listka kielicha kwiatowego, długość (w \(cm\)) płatka kwiatu, szerokość (w \(cm\)) płatka kwiatu. Wykorzystamy teraz algorytm FLD do redukcji wymiaru przestrzeni cech do dwóch. Wizualizacja wyników działania algorytmu FLD na zbiorze Iris została przedstawiona na rysunku 2. Poniżej prezentujemy skrypt w języku python realizujący powyższą procedurę.
import matplotlib.pyplot as plt
from sklearn import datasets
from sklearn.decomposition import PCA
from sklearn.lda import LDA
#importujemy zbiór danych
iris = datasets.load_iris()
X = iris.data #macierz obiektór
y = iris.target #wektor ich poprawnej klasyfikacji
target_names = iris.target_names #nazwa klasy obiektu
#Uruchamiamy algorytm FLD dla 2 komponentów
lda = LDA(n_components=2, solver="eigen")
X_r2 = lda.fit(X, y).transform(X)
#Tworzymy wykres
plt.figure()
for c, i, target_name in zip("rgb", [0, 1, 2], target_names):
plt.scatter(X_r2[y == i, 0], X_r2[y == i, 1], c=c,
label=target_name)
plt.legend()
plt.show()
Metoda składowych głównych (PCA)¶
Jednym z najbardziej znanych algorytmów ekstrakcji cech jest analiza komponentów głównych (ang. Principal Copmonent Analysis (PCA)) (por. Jolliffe [Jolliffe2002], Koronacki [Koronacki2005], Cooley [Cooley1971]). Algorytm ten umożliwia transformację danych z wejściowej przestrzeni cech \(\mathbb{R}^p\) do danych z przestrzeni \(\mathbb{R}^d\) przy czym \(d<p\) tak, by zmienność rzutowanych danych mierzona sumą wariancji była największa. Otrzymane w wyniku działania tej metody nowe cechy są kombinacjami liniowymi oryginalnych cech zbioru uczącego. W metodzie tej zakładamy, że dysponujemy pewnym zbiorem uczącym \(X\), jednak nie musimy posiadać informacji o przynależności klasowej próbek z tego zbioru. Ideą algorytmu PCA jest optymalizacja następującej funkcji celu
przy warunkach
Do optymalizacji powyższej funkcji celu wykorzystujemy następujące twierdzenie
Twierdzenie 1
Niech \(x\) będzie wektorem losowym o wartości oczekiwanej \(\mu\) i macierzy kowariancji \(\Sigma\). Ponadto niech wartości własne \(\lambda_i\) dla \(i=1,\cdots, p\) macierzy \(\Sigma\) będą uporządkowane w porządku malejącym. Wówczas wektor \(t_i\) i-tej składowej głównej wektora \(x\) jest równy i-temu jednostkowemu wektorowi własnemu macierzy kowariancji \(\Sigma\) odpowiadającemu wartości własnej \(\lambda_i\).
Dowód powyższego twierdzenia można znaleźć w [Koronacki2005].
Na podstawie tego twierdzenia zadanie wyznaczenia komponentów głównych sprowadza się do wyznaczenia wektorów własnych macierzy kowariancji \(\Sigma\). Metodę PCA przedstawiamy na poniższym schemacie (algorytm 6).
Algorytm 6 Algorytm PCA
Dane: \(X_{train}\) - zbiór uczący, \(d\) - liczba komponentów
Wyniki: \(T_{PCA}\) - macierz komponentów głównych
- Oblicz macierz kowariancji \(\Sigma\) rozmiaru \(p\times p\).
- Znajdź wartości i wektory własne macierzy kowariancji \(\Sigma\).
- Utwórz macierz komponentów głównych \(T_{PCA}\) rozmiaru \(p\times d\), której kolumny odpowiadają \(d\) największym wartościom własnym macierzy kowariancji \(\Sigma\).
- Oblicz nową macierz \(X_{PCA}\), reprezentującą obiekty w nowej przestrzeni, postaci \(X_{PCA}=X_{train}T_{PCA}\).
Wektor własny odpowiadający \(k\) -tej największej wartości własnej nazywamy \(k\) -tym komponentem głównym. Udowodniono, że kolejne komponenty główne wyznaczają kolejne kierunki największej w sensie wariancji zmienności wektora cech (por. Koronacki [Koronacki2005]). Ponadto, suma wszystkich wartości własnych macierzy kowariancji \(\Sigma\) jest równa sumie wariancji współrzędnych. Aby stwierdzić możliwość redukcji wymiaru przestrzeni cech oraz jej wielkość należy zastosować pewne kryterium. Kryterium to oparte jest na własnościach macierzy symetrycznej. Ponieważ ślad macierzy jest równy sumie wszystkich wartości własnych, zatem suma wszystkich wartości własnych macierzy kowariancji \(\Sigma\) jest równa sumie wariacji poszczególnych współrzędnych. Stąd procent zmienności mierzony wariancją zmierzony przez \(k\) pierwszych komponentów głównych wyraża się wzorem
Ponadto, uzyskana w wyniku zastosowania metody PCA przestrzeń ma następującą cechę. Zastosowana transformacja elementów zbioru uczącego minimalizuje odległości pomiędzy wektorami obserwacji a rzutami, tj.
gdzie \(d(x_i, x_j)\) jest metryką Euklidesa pomiędzy wektorami \(x_i, x_j\), wektory \(x_i^{\star}, x_j^{\star}\) są obrazami wektorów zbioru uczącego \(x_i, x_j\). Ponieważ przy rzutowaniu danych na przestrzeń o niższym wymiarze odległości nie mogą wzrosnąć, w powyższym wzorze można nie uwzględniać modułu różnic odległości.
W praktycznych zastosowaniach jedną z metod ustalania ilości komponentów polega na wybraniu takiej ich liczby aby łącznie wyjaśniały minimum 80% zmienności wariancji.
Przykład 9
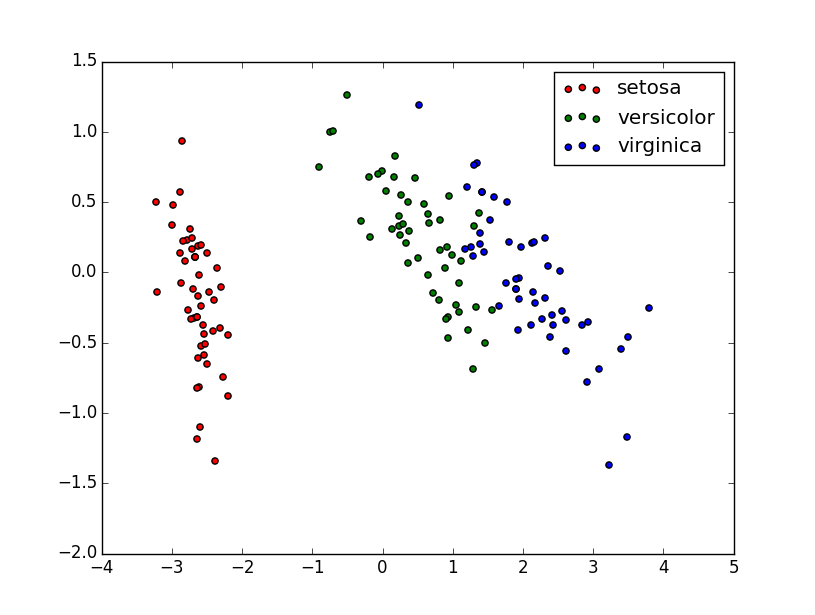
Rozważmy zbiór danych Iris. W zbiorze znajduje się 150 obiektów z trzech różnych gatunków irysów, a każdy z nich jest charakteryzowany przez cztery cechy: długość (w \(cm\).) listka kielicha kwiatowego, szerokość (w \(cm\)) listka kielicha kwiatowego, długość (w \(cm\)) płatka kwiatu, szerokość (w \(cm\)) płatka kwiatu. Wykorzystamy teraz algorytm PCA do redukcji wymiaru przestrzeni cech do dwóch. Pierwszy z komponentów wyjaśnia 92,46% zmienności wariancji, natomiast drugi z nich wyjaśnia 5.3%. Łącznie oba komponenty wyjaśniają 97,76% zmienności wariancji. Poniżej prezentujemy skrypt w języku python realizujący powyższą procedurę.
import matplotlib.pyplot as plt
from sklearn import datasets
from sklearn.decomposition import PCA
from sklearn.lda import LDA
#importujemy zbiór danych
iris = datasets.load_iris()
X = iris.data #macierz obiektór
y = iris.target #wektor ich poprawnej klasyfikacji
target_names = iris.target_names #nazwa klasy obiektu
#Uruchamiamy algorytm PCA dla 2 komponentów
pca = PCA(n_components=2)
X_r = pca.fit(X).transform(X)
#Procent wariancji wyjaśnianej przez metodę dla każdego komponentu
print('Współczynnik wyjaśnianej wariancji dla 2 komponentów: %s'
% str(pca.explained_variance_ratio_))
#Tworzymy wykres
plt.figure()
for c, i, target_name in zip("rgb", [0, 1, 2], target_names):
plt.scatter(X_r[y == i, 0], X_r[y == i, 1], c=c,
label=target_name)
plt.legend()
plt.show()
Dekompozycja na wartości osobliwe (SVD)¶
Inną szeroko stosowaną metodą redukcji wymiaru przestrzeni cech jest dekompozycja na wartości osobliwe (ang. Singular Value decomposition (SVD)) (por. [Gentle1998], [Sarwar2000]).
Definicja 17
Rozkładem macierzy \(X\) rozmiaru \(n\times p\) na wartości osobliwe \(\beta_i\) nazywamy rozkład postaci
gdzie \(S=diag(\beta_1,...,\beta_p)\) jest macierzą wartości osobliwych \(\beta_i\), ponadto \(U^TU=UU^T=I_n\) oraz \(W^TW=WW^T=I_p\). Macierze ortogonalne \(U\) oraz \(W\) tworzą odpowiednio lewe i prawe wektory osobliwe macierzy \(X\), macierz \(I_n\) jest macierzą jednostkową rozmiaru \(n\).
Definicja 18
Wartości \(\beta_i\) nazywamy wartościami osobliwymi.
Twierdzenie 2
Dowolną macierz \(A\) o rozmiarach \(m\times n\) można wyrazić w postaci
gdzie \(S=diag(\beta_1,...,\beta_p)\) jest macierzą wartości osobliwych \(\beta_i\), ponadto \(U^TU=UU^T=I_n\) oraz \(W^TW=WW^T=I_p\). Macierze ortogonalne \(U\) oraz \(W\) tworzą odpowiednio lewe i prawe wektory osobliwe macierzy \(X\).
Dowód znajduje się w [Kincaid2006].
Metoda SVD charakteryzuje się następującymi własnościami. Macierz \(U\) jest macierzą ortogonalną, której kolumny są wektorami własnymi macierzy \(XX^T\). Podobnie macierz \(W\) jest macierzą ortogonalną, której kolumny są wektorami własnymi macierzy \(X^TX\). Jeżeli macierz \(X\) jest macierzą nieosobliwą, to można tak dobrać macierze \(U\) oraz \(W\), żeby jej wszystkie wartości szczególne (osobliwe) były dodatnie. Jeżeli którakolwiek wartość szczególna macierzy \(X\) jest równa \(0\), to macierz ta jest macierzą osobliwą. Wartość bezwzględna wyznacznika kwadratowej macierzy \(X\) jest iloczynem jej wszystkich wartości szczególnych tzn
Metoda Cząstkowych Najmniejszych Kwadratów¶
Metoda Cząstkowych Najmniejszych Kwadratów (ang. Partial Least Squares (PLS)) znajduję liniową zależność pomiędzy dwoma macierzami, zwanymi również blokami, w celu znalezienia tzw. komponentów (zmiennych) ukrytych. Model ten ma postać
gdzie \(T\) jest macierzą komponentów, natomiast \(E\) jest macierzą resztkową. Poniżej przedstawimy ogólne założenia metody PLS. Załóżmy, że mamy macierze \(X\) oraz \(Y\) postaci:
Metoda PLS dokonuje ekstrakcji komponentów ukrytych \(T=[t_1,...,t_k]\). W metodzie tej bierzemy pod uwagę zarówno macierz danych wejściowych \(X\) jak i macierz odpowiedzi \(Y\). Ekstrakcji komponentów dokonujemy w taki sposób, aby kowariancja pomiędzy liniową kombinacją wektorów macierzy \(X\) a macierzą odpowiedzi była jak największa. W metodzie cząstkowych najmniejszych kwadratów maksymalizujemy zatem następującą funkcję celu
przy warunkach
Wektory \(t_k=X_{k-1}w_k\) oraz \(u_k=Y_{k-1}q_k\) nazywamy odpowiednio komponentami macierzy \(X\) oraz \(Y\), natomiast wektory \(w_k\) oraz \(q_k\) nazywamy wektorami wag odpowiednio dla macierzy \(X\) oraz \(Y\). Rysunek 4 przedstawia macierze i wektory występujące w metodzie PLS.
Metoda PLS znajduje zastosowanie w przypadku, kiedy mamy do czynienia z danymi wysoko skorelowanymi, a także z tzw. fat data czyli danymi, gdzie ilość zmiennych znacznie przewyższa ilość próbek. W szczególności metoda ta sprawdza się w zagadnieniach chemometrii, eksperymentach mikromacierzowych, eksperymentach EEG (elektroencyfalogram), ale także w ekonomii oraz badaniach marketingowych. Metoda cząstkowych najmniejszych kwadratów była przedmiotem badań wielu naukowców na przestrzeni ostatnich 40 lat. Poniżej przytaczamy kilka ważnych dat:
- Wold H. (1966, 1975) - Nonlinear Iterative Partial Least Squares (NIPALS) patrz [Wold1966], [Wold1975],
- Wold S. (1983) - PLS1 patrz [Wold1982],
- Höskuldsson (1988) - właściwości PLS [Hoskuldsson1988],
- Helland (1988),
- Rosipal, Trejo (2001) - kernel PLS [Rosipal2001],
- Bastien (2005) - uogólniona liniowa regresja PLS [Bastien2005].
Należy w tym miejscu nadmienić, że metoda PLS jest krytykowana za trudny do interpretacji model. Zrodziło się nawet pytanie czy jest to model statystyczny czy tylko prosty algorytm. Na powyższy model (8) należy raczej patrzeć jako na rodzaj dekompozycji macierzy niż na konkretny model matematyczny. Ponadto krytykę budzi również fakt, że trudno jest zinterpretować deflację macierzy, a także zależności pomiędzy predyktorami obliczone na jej podstawie oraz macierzą odpowiedzi. Próby zbadania aspektów statystycznych metody PLS w jej zastosowaniu do analizy regresji podjęte zostały w pracach Helland [Helland1990], [Helland2001], [Helland2004].
Algorytm NIPALS¶
Algorytm NIPALS został zaproponowany przez H. Wolda w 1966 [Wold1966]. Początkowo algorytm NIPALS służył jako metoda ekstrakcji komponentów macierzy \(X\). Po wyborze pierwszego komponentu był on usuwany z macierzy danych wejściowych przy pomocy techniki deflacji (por. Dodatek matematyczny), po czym algorytm kontynuował pracę wyznaczając drugi komponent dla nowej macierzy danych i tak aż do momentu wyznaczenia wszystkich komponentów. Następnie w 1975 roku [Wold1975] H. Wold zmodyfikował swój algorytm, który od tej pory przy wyznaczaniu komponentów bierze również pod uwagę macierz odpowiedzi. W pracy tej zaproponował on nową wersję algorytmu NIPALS. Algorytm ten przedstawiamy na schemacie (algorytm 7).
Algorytm 7 Algorytm NIPALS
Dane: macierz próbek \(X_0=X\), macierz odpowiedzi \(Y_0=Y\)
Wyniki: model NIPALS
- \(\mathbf{for} \ k=1 \ \mathbf{to} \ d \ \mathbf{do}\)
- ustaw \(u\) na pierwszą kolumnę \(Y\)
- \(\qquad w_k=X^T_{k-1}u_k\)
- \(\qquad t_k=X_{k-1}w_k\)
- \(\qquad q_k=Y_{k-1}^Tt_k\)
- \(\qquad u_k=Y_{k-1}q_k\)
- \(\qquad\) Jeśli nie ma zbieżności to wróć do 3
- \(\qquad p_k=\frac{X^Tt_k}{t_k^Tt_k}\)
- \(\qquad\) Macierz resztowa: \(X_{(k+1)}=X_k-t_kp_k^T\) oraz \(Y_{(k+1)}=Y_k-t_kq_k^T\)
Ideą algorytmu NIPALS jest tzw. dekompozycja biliniowa określona następującymi wzorami
gdzie \(E\) oraz math:F są składnikami resztowymi. Powyższa dekompozycja była szczegółowo badana w monografii Martens, Naes z 1989 (por. [Martens1989]). Dzięki biliniowej dekompozycji otrzymujemy połączenie pomiędzy macierzą math:Y a macierzą math:X za pomocą math:k komponentów math:t_1,…,t_k. Począwszy od algorytmu NIPALS w literaturze pojawiły się różne implementacje metody PLS w jej zastosowaniu do analizy regresji. Oto kilka z nich:
- Wold (1983, 1984) - ortogonalne komponenty macierzy math:X [Wold1982], [Wold1984],
- Martens (1985) - ortogonalne komponenty macierzy math:Y [Martens1985],
- Helland (1988) - [Helland1988],
- de Jong (1993) - SIMPLS [Jong1993].
Regresja PLS - przypadek jednowymiarowy¶
Załóżmy, że macierz odpowiedzi math:Y jest wymiaru math:ntimes 1, tzn.
Metody PLS są z powodzeniem stosowane jako metody klasyfikacji, regresji bądź też jako algorytmy zmniejszające wymiar przestrzeni cech. Metody te stosujemy kiedy dane, z którymi mamy do czynienia są wysoko współliniowe oraz kiedy liczba cech przekracza liczbę próbek. W pracach Geladi, Kowalski [Geladi1986], Tenenhaus [Tenenhaus1998], Wold [Wold2001] można znaleźć ogólne informacje o metodzie PLS. Statystyczne aspekty tej metody opisywane są w pracach Hellanda [Helland1988], [Helland1990], [Helland2001], Garthwaite’a [Garthwaite1994] oraz H”oskuldssona [Hoskuldsson1988], [Hoskuldsson2001]. Metoda PLS często znajduje zastosowanie w takich dziedzinach nauki jak chemometria (dziedzina chemii zajmująca się analizą danych chemicznych, fizycznych, medycznych w celu wyboru optymalnych procedur eksperymentalnych oraz do pozyskania wiedzy o układach chemicznych). Na temat zastosowań PLS w chemometrii istnieje duża liczba publikacji. Wystarczy wspomnieć monografię [Martens1989] czy prace [Wold1982], [Wold2001], [Briandet1996], [Frank1993]. Innym polem zastosowań metody PLS są problemy diagnostyki medycznej takie jak tzw. eksperymenty NIR (ang. Near Infra-Red), czyli eksperymenty oparte na analizie sygnału promieniowania w bliskiej podczerwieni odbitego od powierzchni mózgu, eksperymenty mikromacierzowe zajmujące się analizą genów czy badania EEG (elektroencyfalogram). Również tutaj opublikowano wiele prac, w tym artykuły autorstwa Nguyen, Rocke [Nguyen2002], [Nguyen2002] oraz [Fort2004]. Metody oparte na algorytmie PLS znajdują również zastosowanie w edukacji. Warto tutaj wymienić prace Sellin, Versand [Sellin1995] oraz Noonan [Noonan1979], [Noonan1983]. Ponadto, metody te stosuje się szeroko w ekonomii i badaniach rynku [Fornell1984], [Fornell1994], [Hulland1999]. We wszystkich tych przypadkach cechą wspólną jest duża, a czasami wręcz ogromna, liczba cech w stosunku do małej liczby próbek.
Przykład 11
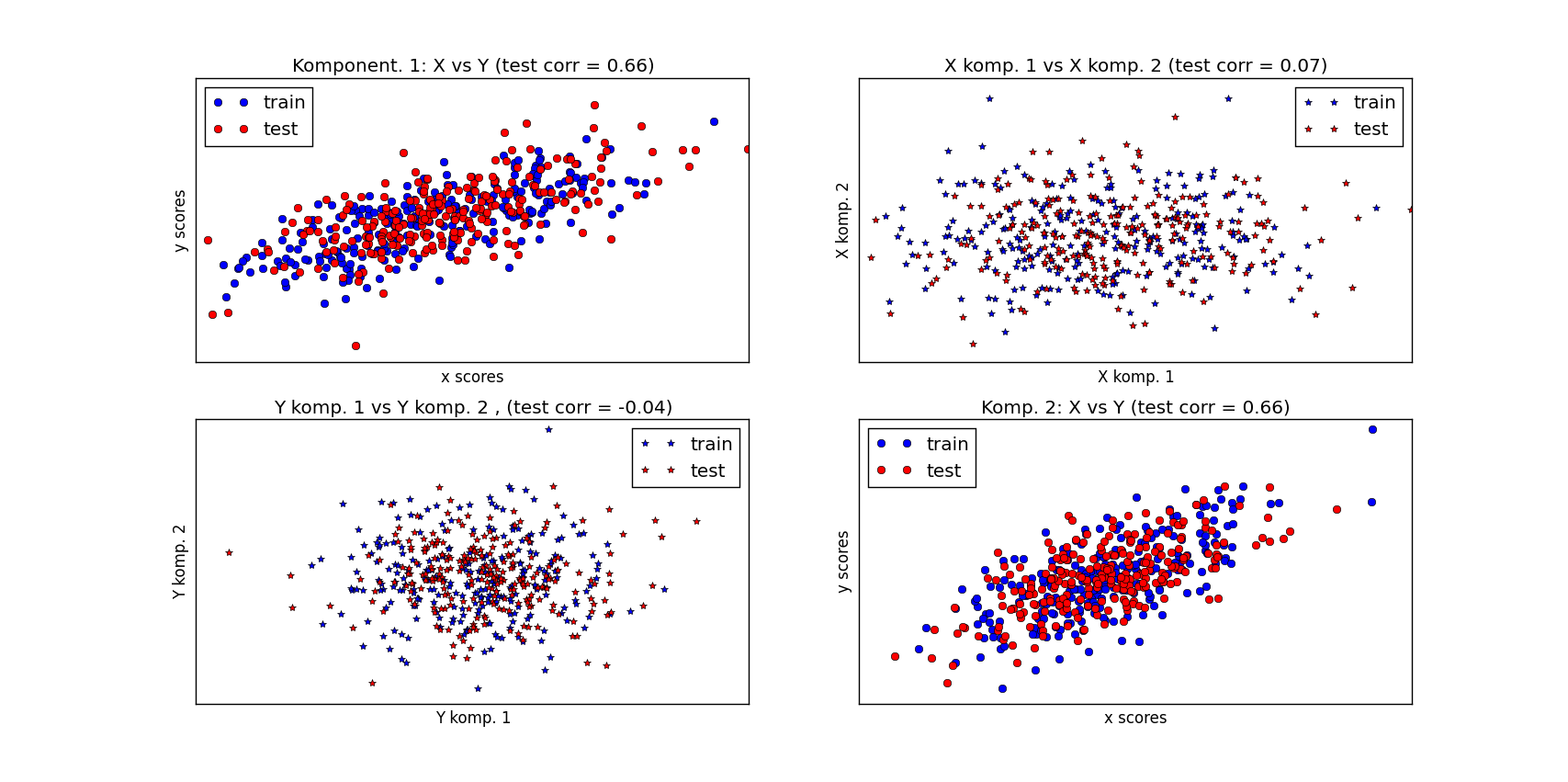
Działanie algorytmu PLS pokażemy na następującym przykładzie. Zbiór danych zawiera \(500\) obiektów charakteryzowanych przez \(4\) cechy i został losowo wygenerowany z rozkładu normalnego. Zbiór ten dzielimy w połowie na część uczącą i testową. Wyznaczamy wektor wag na zbiorze uczącym a następnie dokonujemy redukcji wymiaru przestrzeni cech na zbiorze testowym. Wyniki umieszczamy na wykresie Poniżej prezentujemy skrypt w języku python realizujący powyższą procedurę.
import numpy as np
import pylab as pl
from sklearn.cross_decomposition import PLSCanonical
##########################################################
#Tworzymy zbiór danych
n = 500
#Tworzymy 2 zmienne:
l1 = np.random.normal(size=n)
l2 = np.random.normal(size=n)
latents = np.array([l1, l1, l2, l2]).T
X = latents + np.random.normal(size=4 * n).reshape((n, 4))
Y = latents + np.random.normal(size=4 * n).reshape((n, 4))
#Tworzymy zbiór uczący i testowy
X_train = X[:n / 2]
Y_train = Y[:n / 2]
X_test = X[n / 2:]
Y_test = Y[n / 2:]
print np.shape(Y_train)
print np.shape(X)
print("Corr(X)")
print(np.round(np.corrcoef(X.T), 2))
print("Corr(Y)")
print(np.round(np.corrcoef(Y.T), 2))
#############################################################
# Algorytm PLS
#Wyznaczamy wektor wag
plsca = PLSCanonical(n_components=2)
plsca.fit(X_train, Y_train)
#Redukujemy wymiar przestrzeni cech
X_train_r, Y_train_r = plsca.transform(X_train, Y_train)
X_test_r, Y_test_r = plsca.transform(X_test, Y_test)
#Wyniki umieszczamy na wykresie.
# 1) On diagonal plot X vs Y scores on each components
pl.figure(figsize=(12, 8))
pl.subplot(221)
pl.plot(X_train_r[:, 0], Y_train_r[:, 0], "ob", label="train")
pl.plot(X_test_r[:, 0], Y_test_r[:, 0], "or", label="test")
pl.xlabel("x scores")
pl.ylabel("y scores")
pl.title('Komponent. 1: X vs Y (test corr = %.2f)' %
np.corrcoef(X_test_r[:, 0], Y_test_r[:, 0])[0, 1])
pl.xticks(())
pl.yticks(())
pl.legend(loc="best")
pl.subplot(224)
pl.plot(X_train_r[:, 1], Y_train_r[:, 1], "ob", label="train")
pl.plot(X_test_r[:, 1], Y_test_r[:, 1], "or", label="test")
pl.xlabel("x scores")
pl.ylabel("y scores")
pl.title('Komp. 2: X vs Y (test corr = %.2f)' %
np.corrcoef(X_test_r[:, 1], Y_test_r[:, 1])[0, 1])
pl.xticks(())
pl.yticks(())
pl.legend(loc="best")
# 2) Komponent 1 vs Komponent 2 zbiorów X i Y
pl.subplot(222)
pl.plot(X_train_r[:, 0], X_train_r[:, 1], "*b", label="train")
pl.plot(X_test_r[:, 0], X_test_r[:, 1], "*r", label="test")
pl.xlabel("X komp. 1")
pl.ylabel("X komp. 2")
pl.title('X komp. 1 vs X komp. 2 (test corr = %.2f)'
% np.corrcoef(X_test_r[:, 0], X_test_r[:, 1])[0, 1])
pl.legend(loc="best")
pl.xticks(())
pl.yticks(())
pl.subplot(223)
pl.plot(Y_train_r[:, 0], Y_train_r[:, 1], "*b", label="train")
pl.plot(Y_test_r[:, 0], Y_test_r[:, 1], "*r", label="test")
pl.xlabel("Y komp. 1")
pl.ylabel("Y komp. 2")
pl.title('Y komp. 1 vs Y komp. 2 , (test corr = %.2f)'
% np.corrcoef(Y_test_r[:, 0], Y_test_r[:, 1])[0, 1])
pl.legend(loc="best")
pl.xticks(())
pl.yticks(())
pl.show()
Algorytm PLS1¶
Algorytm PLS1 zaproponowany został przez S. Wolda, syna H. Wolda, w 1983 (por. [Wold1982]). Dokonuje on ekstrakcji wektora wag \(w_k\) oraz komponentów \(t_k\), a następnie oblicza wektory \(p_k=X_{k-1}^Tt_k\) oraz \(q_k=Y^Tt_k\). Takie podejście pozwala na wykonanie biliniowej dekompozycji danej wzorami (9) oraz (10). Algorytm PLS1 przedstawiamy na schemacie 8.
Algorytm 8 Algorytm PLS1
Dane: macierz próbek \(X_0=X\), macierz odpowiedzi \(Y_0=Y\)
Wyniki: model PLS1
- \(\mathbf{for} \ k=1 \ \mathbf{to} \ d \ \mathbf{do}\)
- \(\qquad w_k=\frac{X^T_{k-1}Y_{k-1}}{\|X^T_{k-1}Y_{k-1}\|}\)
- \(\qquad t_k=X_{k-1}w_k\)
- \(\qquad p_k=\frac{X^Tt_k}{t_k^Tt_k}\)
- \(\qquad q_k=\frac{Y_{k-1}^Tt_k}{t_k^Tt_k}\)
- \(\qquad\) Macierz resztowa: \(X_{(k+1)}=X_k-t_kp_k^T\) oraz \(Y_{(k+1)}=Y_k-t_kq_k^T\)
Zauważmy, że wartości \(q_n\) wymiaru \(n\times 1\) są współczynnikami regresji \(t_k\) na \(Y\). Macierz wszystkich współczynników regresji uzyskanych w \(k\) iteracjach oznaczamy symbolem \(\hat{q}_k=(q_1,...,q_k)\). Współczynniki te uzyskujemy metodą najmniejszych kwadratów. Następnie wektor \(\hat{q}_k\) transformujemy z powrotem do oryginalnych zmiennych, dzięki czemu otrzymujemy wektor współczynników regresji wymiaru \(p\times 1\). Wektor ten oznaczamy poprzez \(B_k^{PLS}\). Współczynniki te są dane wzorem
gdzie \(W=(w_1,\cdots,w_k)\), \(P=(p_1,\cdots,p_k)\), oraz \(\hat{q}\) silnie zależą od macierzy odpowiedzi \(Y\). Stopień złożoności metody PLS jest kontrolowany poprzez liczbę komponentów \(t_k\) w modelu regresji. Wybór wartości \(k\) ma wpływ nie tylko na złożoność modelu lecz również na jego sprawność.
Twierdzenie 3
Dla wektorów wag oraz komponentów uzyskanych metodą PLS zachodzą następujące własności:
- \(t_k\bot t_j\) dla \(k\not=j\),
- \(w_k\bot w_j\) dla \(k\not=j\),
- \(t_k^TX_l=0\) dla \(l>k\),
- \(Y^T_{k-1}t_k=Y_0^Tt_k\).
Dowód znajduje się w pracy Höskuldsson [Hoskuldsson1988].
Algorytm ten możemy również zapisać za pomocą narzędzi statystycznych w sposób, który przedstawiany na schemacie (algorytm 9).
Algorytm 9 Regresja PLS
Dane: macierz próbek \(X_0=X\), macierz odpowiedzi \(Y_0=Y\)
Wyniki:
- \(\mathbf{for} \ k=1 \ \mathbf{to} \ d \ \mathbf{do}\)
- \(\qquad\) Oblicz:math:w_k.
- \(\qquad w_k=\mathop{\rm argmax}_{w_k}\{\mathop{\rm Cov}(X_{k-1}w_k,Y_{k-1})\}\).
- \(\qquad t_k=X_{k-1}w_k\).
- \(\qquad\) Normalizuj każde \(x_{j,k-1}\) poprzez \(t_k\).
- \(\qquad\) Normalizuj każde \(y_{k-1}\) poprzez \(t_k\).